ការពិសោធន៍ RT-qPCR រួមមានការទាញយក RNA និងការវាយតម្លៃគុណភាព ការចម្លងបញ្ច្រាស និង qPCR បីជំហាន ដែលជំហាននីមួយៗមានការប្រុងប្រយ័ត្នជាច្រើន យើងនឹងណែនាំលម្អិតខាងក្រោម។
Ⅰការវាយតម្លៃគុណភាព RNA
នៅក្នុងការពិសោធន៍ RT-qPCR បន្ទាប់ពីការបញ្ចប់នៃការទាញយក RNA គុណភាពនៃ RNA ចាំបាច់ត្រូវវាយតម្លៃ ហើយការពិសោធន៍តាមដានអាចត្រូវបានអនុវត្តបន្ទាប់ពីវាមានលក្ខណៈគ្រប់គ្រាន់។វិធីសាស្រ្តវាយតម្លៃរួមមាន spectrophotometer, Agilent gel electrophoresis, Agilent 2100 analysis, ក្នុងចំណោមនោះ spectrophotometer ដែលប្រើជាទូទៅបំផុត និងការរកឃើញវិធីសាស្រ្ត electrophoresis gel agarose ។វាគួរតែត្រូវបានកត់សម្គាល់ថាវិធីសាស្រ្តទាំងពីរនេះចាំបាច់ត្រូវប្រើរួមគ្នាដើម្បីបញ្ចប់ការរកឃើញនិងការវិភាគនៃកំហាប់ RNA ភាពបរិសុទ្ធនិងភាពសុចរិតដើម្បីធានាគុណភាពនៃ RNA ។
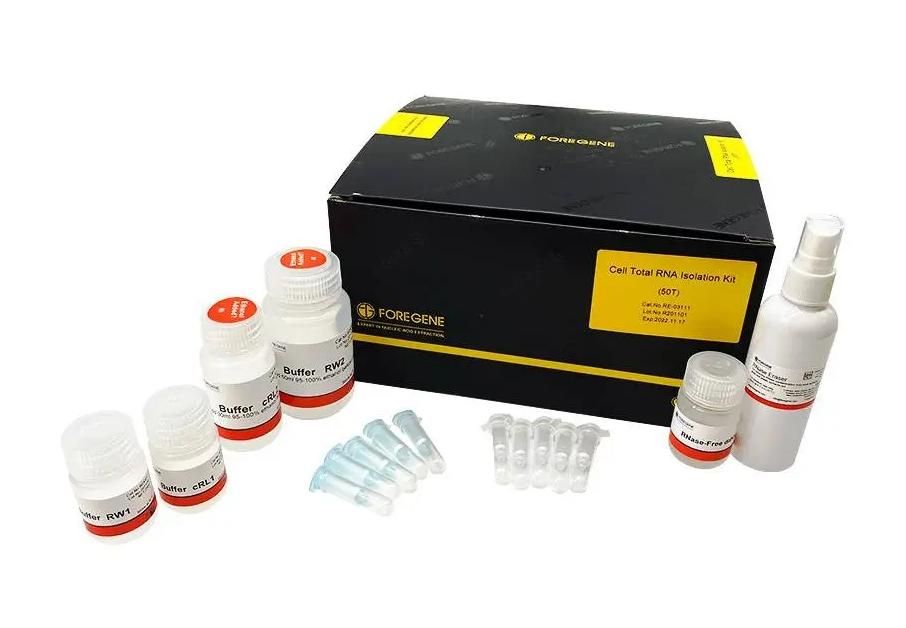
ឧបករណ៍ញែក RNA ពាក់ព័ន្ធ៖
សំណុំកោសិកាដាច់ពីគ្នា RNA សរុប
RNA សរុបដែលបន្សុត និងគុណភាពខ្ពស់អាចទទួលបានពីកោសិកាវប្បធម៌ផ្សេងៗក្នុងរយៈពេល 11 នាទី។
ទាញយក RNA សរុបដែលមានភាពបរិសុទ្ធ និងគុណភាពខ្ពស់យ៉ាងរហ័ស និងមានប្រសិទ្ធភាពពីជាលិកាសត្វផ្សេងៗ។
ឧបករណ៍វាស់ស្ទង់វិសាលគម៖
spectrophotometer ត្រូវបានប្រើជាចម្បងដើម្បីកំណត់កំហាប់ និងភាពបរិសុទ្ធនៃ RNA ប៉ុន្តែវាមិនអាចរកឃើញភាពសុចរិតនៃ RNA និងសំណល់ហ្សែនបានទេ។ក្នុងចំណោមពួកគេ A260/280 និង A260/230 គឺជាប៉ារ៉ាម៉ែត្រសំខាន់សម្រាប់ការរកឃើញភាពបរិសុទ្ធ RNA ហើយភាពបរិសុទ្ធ RNA អាចត្រូវបានរកឃើញដោយយោងទៅតាមការប្រែប្រួលនៃតម្លៃរបស់វា៖
1. 1.9< A260/280< 2.1 ដែលបង្ហាញថាភាពបរិសុទ្ធ RNA គឺល្អA260/280<1.9 ដែលបង្ហាញថាអាចមានសំណល់ប្រូតេអ៊ីននៅក្នុង RNA;A260/280>2.1 ដែលបង្ហាញពីការរិចរិលផ្នែកខ្លះនៃ RNA ដែលអាចត្រូវបានបញ្ជាក់បន្ថែមដោយ electrophoresis ជែល agarose ។
2. 2.0< A260/230< 2.2 ដែលបង្ហាញថាភាពបរិសុទ្ធ RNA គឺល្អA260/230< 2.0 ដែលបង្ហាញថាអាចមានសំណល់នៃសារធាតុសរីរាង្គនៅក្នុង RNA ដូចជា phenols អេតាណុល ឬជាតិស្ករ។
Agarose gel electrophoresis៖
Agarose gel electrophoresis assay អាចវិភាគភាពសុចរិតរបស់ RNA, ហ្សែន និងសំណល់ប្រូតេអ៊ីន ប៉ុន្តែមិនអាចកំណត់បរិមាណកំហាប់ RNA ឱ្យបានត្រឹមត្រូវ ឬរកឃើញសំណល់នៃសារធាតុសរីរាង្គនោះទេ។យកគំរូ eukaryotic RNA ជាឧទាហរណ៍៖
1. RNA ត្រូវបានទទួលរងនូវ agarose gel electrophoresis ។ប្រសិនបើមានតែបីក្រុមនៃ 28sRNA, 18sRNA និង 5.8sRNA នៅលើផែនទីជែល វាបង្ហាញថា RNA ដែលបានស្រង់ចេញគឺនៅដដែល។ប្រសិនបើមានបាតុភូតអូសវាបង្ហាញពីការរិចរិលដោយផ្នែកនៃ RNA ។
2. ប្រសិនបើមានក្រុមភ្លឺតែមួយរវាងរន្ធកាវ និងក្រុមតន្រ្តី 28sRNA វាអាចមានសំណល់ DNA ហ្សែន។
3. ប្រសិនបើដុំពកលេចឡើងក្នុងរន្ធកាវបិទ វាបង្ហាញថាអាចមានសំណល់នៃប្រូតេអ៊ីន និងសារធាតុម៉ាក្រូម៉ូលេគុលផ្សេងទៀត។
Ⅱ. ប្រតិចារិកបញ្ច្រាស
បន្ទាប់ពីការទាញយក RNA ត្រូវបានបញ្ចប់ វាចាំបាច់ត្រូវប្តូរទៅជា cDNA សម្រាប់ការពិសោធន៍ជាបន្តបន្ទាប់ ដូច្នេះជំហានបញ្ច្រាសគឺចាំបាច់។ប្រតិចារិកបញ្ច្រាសនឹងត្រូវបានណែនាំពីការជ្រើសរើសនៃការចម្លងបញ្ច្រាស និងបឋម៖
ការជ្រើសរើស ប្រតិចារិកបញ្ច្រាស៖
ប្រតិចារិកបញ្ច្រាសធម្មតារួមមាន AMV RTase និង MMLV RTase ។RNase H នៃ AMV RTase មានសកម្មភាពខ្លាំង ប្រវែងសំយោគខ្លី បរិមាណសំយោគទាប និងស្ថេរភាពកម្ដៅល្អ (42 ~ 55 ℃) ។សកម្មភាព RNase H នៃ MMLV RTase គឺខ្សោយ ប្រវែងសំយោគវែង បរិមាណសំយោគខ្ពស់ ហើយស្ថេរភាពកម្ដៅខ្សោយ (37 ~ 42 ℃) ។
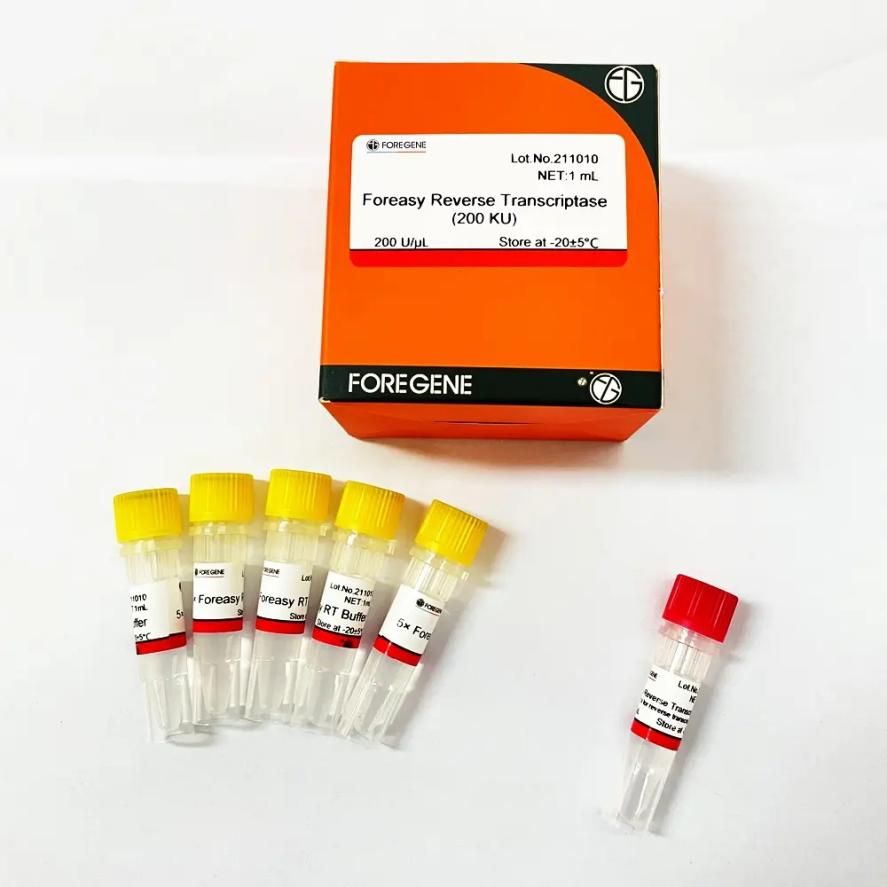
ដោយសារតែអង់ស៊ីម RNase H មានមុខងារនៃការបំប្លែងគំរូ RNA នោះ MMLV ដែលមានសកម្មភាព RNase H ខ្សោយគួរតែត្រូវបានជ្រើសរើសជាអាទិភាពក្នុងអំឡុងពេលប្រតិចារិកបញ្ច្រាស ហើយបន្ទាប់ពីវិស្វកម្មហ្សែនក្រោយៗមក ស្ថេរភាពកម្ដៅនៃ MMLV បានឈានដល់កម្រិតមួយប្រកបដោយគុណភាព។លេបថ្នាំ ForegeneForeasy Reverse Transcriptase (M-MLV សម្រាប់ការចម្លងបញ្ច្រាស) ជាឧទាហរណ៍ វាគឺជា transcriptase បញ្ច្រាសថ្មីដែលបានបង្ហាញនៅក្នុងបាក់តេរី E. coli ដែលត្រូវបានកែច្នៃដោយប្រើប្រាស់បច្ចេកវិទ្យា genetic recombination technology។វាគឺជា DNA polymerase ដែលផ្សំឡើងវិញដែលសំយោគខ្សែ DNA បន្ថែមពីខ្សែតែមួយ RNA, DNA ឬ RNA:DNA hybrid ។វាមិនមានសកម្មភាព RNase H ស្ថេរភាពខ្លាំង ភាពស្និទ្ធស្នាលរបស់ RNA ខ្លាំង និងភាពរសើបនៃការរកឃើញខ្ពស់។
Foreasy Reverse Transcriptase (M-MLV សម្រាប់ការចម្លងបញ្ច្រាស)
ការជ្រើសរើស primer៖
ជាទូទៅថ្នាំ primers RT ចែកចេញជាបីប្រភេទ៖ oligo dT, random primers និង gene-specific primers។ជ្រើសរើសថ្នាំ primers សមរម្យសម្រាប់ប្រើតាមតម្រូវការពិសោធន៍ផ្សេងៗគ្នា។
1. ប្រសិនបើគំរូមានប្រភពដើម eukaryotic ហើយ cDNA ចុងត្រូវបានប្រើសម្រាប់ការពង្រីក PCR ជាប្រចាំនោះ Oligo (dT) ត្រូវបានណែនាំ។ប្រសិនបើការពិសោធន៍ជាបន្តបន្ទាប់ត្រូវបានប្រើសម្រាប់តែ qPCR នោះ Oligo (dT) ត្រូវបានណែនាំអោយលាយជាមួយនឹង primers ចៃដន្យ ដើម្បីបង្កើនប្រសិទ្ធភាពនៃការចម្លងបញ្ច្រាស។
2. ប្រសិនបើគំរូគឺមកពី prokaryotes នោះ Random Primers ឬ gene specific primers គួរតែត្រូវបានជ្រើសរើសសម្រាប់ការចម្លងបញ្ច្រាស។
Ⅲ.qPCR
បរិមាណ fluorescence ត្រូវបានពិពណ៌នាជាចម្បងពីការជ្រើសរើសវិធីសាស្រ្តបរិមាណ គោលការណ៍រចនាបឋម ការជ្រើសរើស ROX ការកំណត់រចនាសម្ព័ន្ធប្រព័ន្ធប្រតិកម្ម និងការកំណត់លក្ខខណ្ឌប្រតិកម្ម។ល។
ការជ្រើសរើសវិធីសាស្ត្របរិមាណ៖
វិធីសាស្រ្តបរិមាណត្រូវបានបែងចែកទៅជាវិធីសាស្រ្តបរិមាណទាក់ទង និងវិធីសាស្រ្តបរិមាណដាច់ខាត។ការវិភាគបរិមាណដែលទាក់ទងអាចត្រូវបានប្រើដើម្បីរកមើលឥទ្ធិពលនៃវិធីសាស្ត្រព្យាបាលជាក់លាក់លើការបញ្ចេញហ្សែន រកឃើញភាពខុសគ្នានៃការបញ្ចេញហ្សែននៅពេលផ្សេងៗគ្នា និងប្រៀបធៀបភាពខុសគ្នានៃការបញ្ចេញហ្សែននៅក្នុងជាលិកាផ្សេងៗ។បរិមាណដាច់ខាតអាចរកឃើញបរិមាណអាស៊ីតនុយក្លេអ៊ីកនៅក្នុងមេរោគជាដើម។នៅពេលធ្វើការពិសោធន៍ យើងត្រូវជ្រើសរើសវិធីសាស្ត្របរិមាណសមស្របទៅតាមការពិសោធន៍របស់យើងផ្ទាល់។
គោលការណ៍នៃការរចនាបឋម៖
ការរចនានៃ primer សម្រាប់ qPCR គឺទាក់ទងដោយផ្ទាល់ទៅនឹងប្រសិទ្ធភាព amplification និងភាពជាក់លាក់នៃផលិតផល។ដូច្នេះ ការរចនា primers ល្អត្រឹមត្រូវ គឺជាជំហានដំបូងនៃ qPCR ជោគជ័យ។នៅក្នុងការរចនានៃ primer គោលការណ៍ដូចខាងក្រោមគួរតែត្រូវបានយកចិត្តទុកដាក់នៅពេលបំពេញតាមគោលការណ៍នៃការរចនា primer សាមញ្ញ:
1. ប្រវែងនៃបំណែកគោលដៅត្រូវបានគ្រប់គ្រងចន្លោះពី 100 ទៅ 300 bp;
2. ការរចនា Cross-exon ដើម្បីជៀសវាងឥទ្ធិពលនៃ DNA ហ្សែន;
3. primers ដែលបានរចនាឡើងត្រូវតែត្រូវបានសាកល្បងសម្រាប់ប្រសិទ្ធភាព amplification ហើយលុះត្រាតែប្រសិទ្ធភាព amplification ឈានដល់ស្តង់ដារ (90-110%) ពួកគេអាចប្រើសម្រាប់ការពិសោធន៍បរិមាណ។
4. ការផ្តោតអារម្មណ៍បឋមជាធម្មតាត្រូវបានធ្វើឱ្យប្រសើររវាង 0.1uM និង 1.0uM ។
ការជ្រើសរើសROX៖
នៅក្នុងដំណើរការនៃប្រតិកម្មបរិមាណ ROX អាចកែតម្រូវភាពខុសគ្នានៃផ្លូវអុបទិក កំហុសក្នុងការដាក់បំពង់ ឬភាពខុសគ្នានៃបរិមាណដែលបណ្តាលមកពីការហួត និង condensation ស្មើភាពគ្នា ធ្វើអោយប្រសើរឡើងនូវភាពអាចឡើងវិញនៃលទ្ធផល។ទោះជាយ៉ាងណាក៏ដោយគួរកត់សំគាល់ថាការជ្រើសរើស ROX គឺទាក់ទងនឹងឧបករណ៍។ប្រសិនបើឧបករណ៍ qPCR មានមុខងារកែតម្រូវដោយស្វ័យប្រវត្តិនូវភាពខុសគ្នារវាងរន្ធ វាមិនចាំបាច់បន្ថែម ROX ទេ។បើមិនដូច្នោះទេវាត្រូវបន្ថែមការកែតម្រូវ ROX ។ដៃគូតូចៗក្នុងការទិញសារធាតុប្រតិកម្មត្រូវតែស្របតាមឧបករណ៍ដែលប្រើដើម្បីជ្រើសរើស ROX ត្រឹមត្រូវ ជៀសវាងកំហុសនៅពេលក្រោយ។
ការរៀបចំប្រព័ន្ធប្រតិកម្ម៖
បរិមាណប្រតិកម្ម 20ul និង 50ul ត្រូវបានគេពេញចិត្ត។បញ្ហាខាងក្រោមគួរតែត្រូវបានយកចិត្តទុកដាក់នៅពេលដែលប្រព័ន្ធត្រូវបានបង្កើតឡើង:
1. ប្រព័ន្ធប្រតិកម្មត្រូវរៀបចំដោយខ្យល់ចេញចូលនៅក្នុងកន្លែងធ្វើការដែលស្អាតខ្លាំង ឌីអេច ថ្មី2O ត្រូវបានប្រើសម្រាប់ការពិសោធន៍នីមួយៗ;
2. ការពិសោធន៍នីមួយៗត្រូវរៀបចំ NTC ដើម្បីផ្ទៀងផ្ទាត់ថាតើមានការបំពុលនៅក្នុងប្រព័ន្ធ ហើយរាល់គូនៃ primers ត្រូវធ្វើ NTC នៅពេលរៀបចំប្រព័ន្ធ។
3. ដើម្បីរកមើលថាតើមានសំណល់ gDNA នៅក្នុងគំរូ RNA ដែរឬទេ NRT អាចត្រូវបានរៀបចំសម្រាប់គំរូនីមួយៗសម្រាប់ការរកឃើញ។
4. នៅពេលរៀបចំប្រព័ន្ធ វាត្រូវបានណែនាំឱ្យធ្វើម្តងទៀតនូវបច្ចេកទេសយ៉ាងហោចណាស់ 3 ដងសម្រាប់គំរូមួយ។
5. នៅពេលដែលគំរូគឺ cDNA វាត្រូវបានផ្ដល់អនុសាសន៍ឱ្យពនឺ 5-10 ដងដើម្បីកាត់បន្ថយឥទ្ធិពលរារាំងនៃប្រព័ន្ធចម្លងបញ្ច្រាសនៅលើការពិសោធន៍ qPCR ។វាជាការប្រសើរក្នុងការស្វែងយល់ពីបរិមាណគំរូដោយជម្រាល ដូច្នេះតម្លៃ CT ធ្លាក់ចន្លោះពី 20-30;
6. កំណត់ចំនួនប្រតិកម្មដែលត្រូវការ, កើនឡើង 5-10% នៅលើមូលដ្ឋាននៃចំនួននៃប្រតិកម្ម, និងគណនាលេខកំណត់រចនាសម្ព័ន្ធកម្រិតសំឡេង;
7, ប្រព័ន្ធត្រូវបានរៀបចំដោយប្រើគោលការណ៍ premix, លាយបន្ទាប់ពីការ centrifugation និងធានាថាមិនមានពពុះ;
8, តាមដែលអាចធ្វើបានដើម្បីជ្រើសរើសសម្ភារៈប្រើប្រាស់។
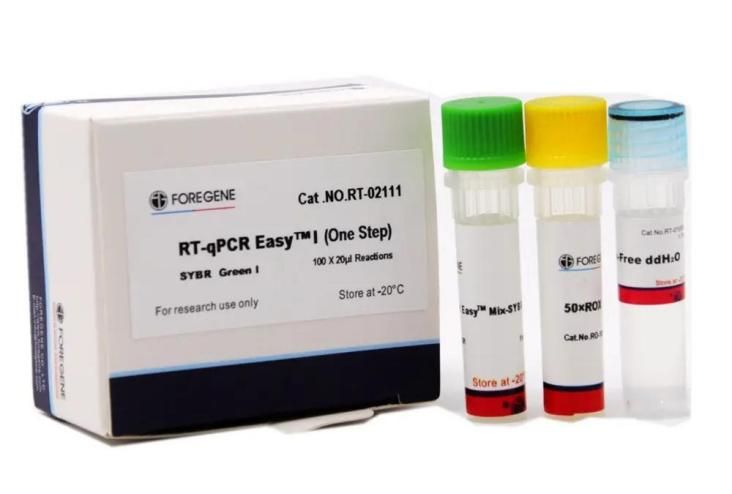
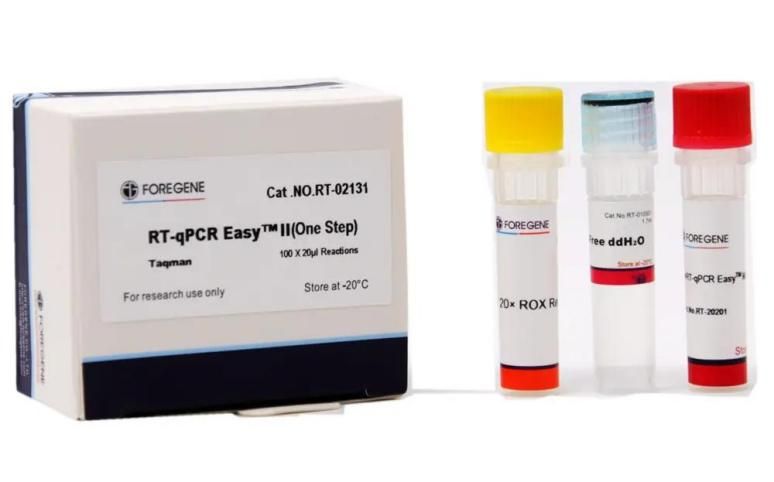
កញ្ចប់ RT-qPCR ពាក់ព័ន្ធ
កញ្ចប់នេះប្រើសារធាតុចម្លងបញ្ច្រាស Foregene តែមួយគត់ និង Foregene HotStar Taq DNA Polymerase រួមបញ្ចូលគ្នាជាមួយប្រព័ន្ធប្រតិកម្មពិសេសមួយ ដើម្បីបង្កើនប្រសិទ្ធភាពនៃការពង្រីក និងភាពជាក់លាក់នៃប្រតិកម្ម។
ពេលវេលាប្រកាស៖ ថ្ងៃទី ២៣ ខែមេសា ឆ្នាំ ២០២៣