ភាពជាក់លាក់នៃការរកឃើញ
ក្នុងករណីភាគច្រើន គោលបំណងនៃការរចនាបឋមគឺដើម្បីបង្កើនភាពជាក់លាក់នៃ PCR ។នេះត្រូវបានកំណត់ដោយឥទ្ធិពលដែលអាចព្យាករណ៍បានច្រើន ឬតិចនៃអថេរជាច្រើន។អថេរសំខាន់មួយគឺលំដាប់នៅខាងចុង 3′ នៃ primer ។
សំខាន់ ការវិភាគ PCR ដែលត្រូវបានរចនាឡើងសម្រាប់ភាពជាក់លាក់ទំនងជាអាចរក្សាបាននូវប្រសិទ្ធភាពខ្ពស់លើជួរថាមវន្តដ៏ធំទូលាយមួយ ពីព្រោះការវិភាគមិនផលិតផលិតផលពង្រីកដែលមិនជាក់លាក់ ដោយហេតុនេះប្រកួតប្រជែងជាមួយថ្នាំ PCR ឬរារាំងប្រតិកម្ម amplification សំខាន់។
ជាការពិតណាស់ ក្នុងករណីខ្លះ ភាពជាក់លាក់មិនមែនជារឿងសំខាន់បំផុតនោះទេ ជាឧទាហរណ៍ នៅពេលដែលគោលដៅគឺដើម្បីកំណត់បរិមាណដែលទាក់ទងយ៉ាងជិតស្និទ្ធ ប៉ុន្តែធាតុបង្កជំងឺផ្សេងៗគ្នា ការរចនាពិសេស ការបង្កើនប្រសិទ្ធភាព និងស្តង់ដារផ្ទៀងផ្ទាត់ត្រូវបានទាមទារ។
ខ្សែកោងរលាយគឺជាវិធីសាស្រ្តស្តង់ដារសម្រាប់វាយតម្លៃភាពជាក់លាក់នៃ amplicons យ៉ាងហោចណាស់ក្នុងលក្ខខណ្ឌថាតើត្រូវពង្រីកគោលដៅតែមួយ។ទោះជាយ៉ាងណាក៏ដោយវាត្រូវតែត្រូវបានសង្កត់ធ្ងន់ថាខ្សែកោងរលាយអាចមានការយល់ច្រឡំព្រោះឧទាហរណ៍ពួកវាអាចត្រូវបានប៉ះពាល់ដោយឥទ្ធិពលរួមបញ្ចូលគ្នានៃ primers ល្អបំផុតនិងការប្រមូលផ្តុំគំរូទាប។
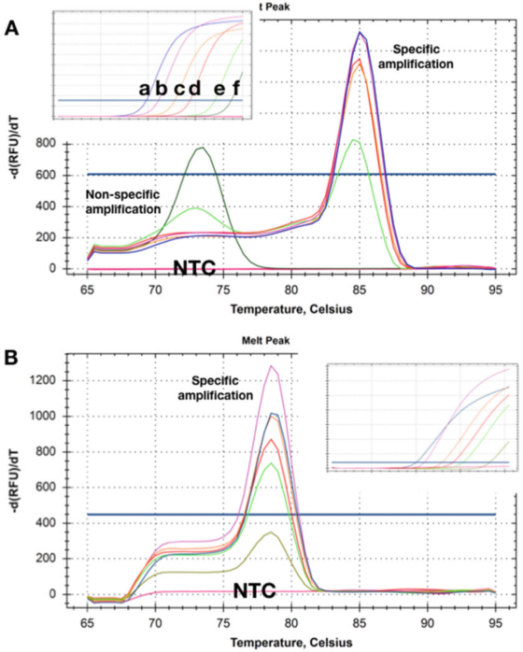
P៥ |ខ្សែកោងរលាយបង្ហាញពីការផ្លាស់ប្តូរ Tm ដែលទទួលបានពីការរកឃើញពីរនៃបរិមាណផ្សេងគ្នានៃ DNA គោលដៅពីរ។
ក. នៅកំហាប់ខ្ពស់ (ផ្សាយពាណិជ្ជកម្ម)) មិនមានសារធាតុ primer dimer ជាក់ស្តែងបន្ទាប់ពីការវាស់វែង qPCR ត្រូវបានបញ្ចប់។នៅពេលដែលកំហាប់គំរូថយចុះដល់ 50 ច្បាប់ចម្លង (e) ផលិតផលដែលមិនជាក់លាក់ចាប់ផ្តើមលេចឡើង ហើយក្លាយជាផលិតផលតែមួយគត់ដែលមានកំហាប់ទាបបំផុត (f) ។
ខ. ការធ្វើតេស្តបានកត់ត្រា Tms ដូចគ្នានៅកំហាប់គោលដៅទាំងអស់ ហើយមិនមាន primer dimer ជាក់ស្តែងសូម្បីតែនៅកំហាប់ទាបបំផុត (5 ច្បាប់ចម្លង)។នៅពេលប្រើវិធីសាស្រ្តរាវរកទាំងពីរនេះ គ្មានផលិតផល amplification ត្រូវបានរកឃើញនៅក្នុង NTCs ទេ។
P5 បង្ហាញពីខ្សែកោងរលាយដែលទទួលបានជាមួយនឹងគំរូដែលគំរូមានវត្តមាននៅកំហាប់ផ្សេងៗគ្នា។P 5a បង្ហាញថានៅកំហាប់ទាបបំផុតពីរ Tms នៃផលិតផល amplification មិនជាក់លាក់ដែលផលិតគឺទាបជាងអំពែរជាក់លាក់។
ជាក់ស្តែង វិធីសាស្ត្ររាវរកនេះមិនអាចត្រូវបានប្រើប្រាស់យ៉ាងជឿជាក់ដើម្បីស្វែងរកគោលដៅដែលមាននៅក្នុងកំហាប់ទាបនោះទេ។
គួរឱ្យចាប់អារម្មណ៍ NTCs ពោលគឺសំណាកដែលគ្មាន DNA ទាល់តែសោះ មិនបានកត់ត្រាផលិតផលពង្រីក (មិនជាក់លាក់) ដោយបង្ហាញថា DNA ហ្សែនផ្ទៃខាងក្រោយអាចចូលរួមក្នុងការពង្រីក/វត្ថុធាតុ polymerization មិនជាក់លាក់។
ពេលខ្លះការពង្រឹងផ្ទៃខាងក្រោយ និងការពង្រីកមិនជាក់លាក់បែបនេះមិនអាចដោះស្រាយបានទេ ប៉ុន្តែជារឿយៗវាអាចទៅរួចក្នុងការរចនាវិធីសាស្ត្ររាវរកដែលមិនមានការពង្រីកមិនជាក់លាក់នៅក្នុងការប្រមូលផ្តុំគំរូណាមួយ និង NTC (P 5b) ។
នៅទីនេះសូម្បីតែការកត់ត្រាការពង្រីកនៃការផ្តោតអារម្មណ៍គោលដៅជាមួយនឹង Cq នៃ 35 នឹងបង្កើតខ្សែកោងរំលាយជាក់លាក់មួយ។ដូចគ្នានេះដែរ NTCs មិនបានបង្ហាញសញ្ញានៃការពង្រីកមិនជាក់លាក់ទេ។ពេលខ្លះ ឥរិយាបថរាវរកអាចពឹងផ្អែកលើស្រាម្តាយ ហើយមានតែការពង្រីកមិនជាក់លាក់ប៉ុណ្ណោះដែលត្រូវបានរកឃើញនៅក្នុងសមាសធាតុសតិបណ្ដោះអាសន្នមួយចំនួន ដែលអាចទាក់ទងនឹងកំហាប់ Mg2+ ផ្សេងៗគ្នា។
ស្ថេរភាពនៃការរកឃើញ
ការបង្កើនប្រសិទ្ធភាពនៃ Ta គឺជាជំហានដ៏មានប្រយោជន៍ក្នុងដំណើរការផ្ទៀងផ្ទាត់ និងបង្កើនប្រសិទ្ធភាពនៃការរកឃើញ qPCR ។វាផ្តល់នូវការចង្អុលបង្ហាញដោយផ្ទាល់អំពីភាពរឹងមាំនៃ primer ដែលបានកំណត់ដោយបង្ហាញសីតុណ្ហភាព (ឬជួរសីតុណ្ហភាព) ដែលផលិត Cq ទាបបំផុតដោយមិនពង្រីក NTC ។
ភាពខុសគ្នាពី 2 ទៅ 4 ដងនៃភាពប្រែប្រួលអាចមិនសំខាន់សម្រាប់អ្នកដែលមានការបញ្ចេញមតិ mRNA ខ្ពស់ ប៉ុន្តែសម្រាប់ការធ្វើតេស្តរោគវិនិច្ឆ័យ វាអាចមានន័យថាភាពខុសគ្នារវាងលទ្ធផលវិជ្ជមាន និងអវិជ្ជមានមិនពិត។
លក្ខណៈសម្បត្តិ Ta នៃ primers qPCR អាចប្រែប្រួលយ៉ាងខ្លាំង។ការធ្វើតេស្តមួយចំនួនមិនមានភាពរឹងមាំខ្លាំងទេ ហើយប្រសិនបើពួកគេមិនត្រូវបានអនុវត្តក្រោមតម្លៃ Ta ល្អបំផុតនៃ primers នោះពួកគេនឹងដួលរលំយ៉ាងឆាប់រហ័ស។
នេះមានសារៈសំខាន់ ដោយសារការរកឃើញប្រភេទនេះច្រើនតែមានបញ្ហានៅក្នុងពិភពពិត ហើយភាពបរិសុទ្ធនៃគំរូ ការប្រមូលផ្តុំ DNA ឬវត្តមាន DNA ផ្សេងទៀតប្រហែលជាមិនល្អបំផុតនោះទេ។
លើសពីនេះ លេខចម្លងគោលដៅអាចប្រែប្រួលក្នុងជួរដ៏ធំទូលាយមួយ ហើយសារធាតុប្រតិកម្ម ប្រដាប់ប្រើប្រាស់ផ្លាស្ទិច ឬឧបករណ៍អាចមានភាពខុសប្លែកពីឧបករណ៍ដែលបានប្រើនៅពេលដំឡើងការធ្វើតេស្ត។
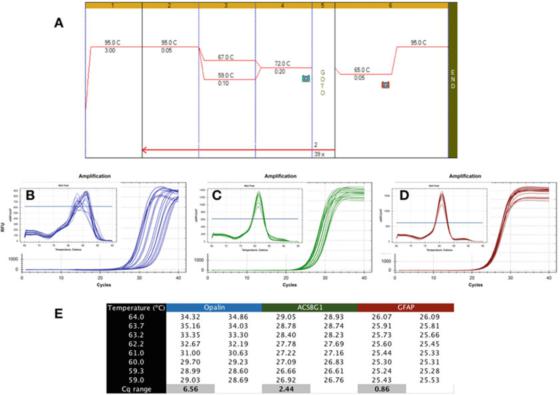
P6|ជម្រាលសីតុណ្ហភាពបង្ហាញពីភាពរឹងមាំផ្សេងគ្នានៃការរកឃើញ PCR ។
A. ប្រើ Sensifast SYBR mastermix របស់ Bioline (លេខកាតាឡុក BIO-98050) ដើម្បីអនុវត្ត PCR លើ cDNA ដែលរៀបចំពី RNA ខួរក្បាលរបស់មនុស្ស។
ខ. ប្រើឧបករណ៍ CFX qPCR របស់ Bio-Rad ដើម្បីកត់ត្រាផែនទីពង្រីក និងខ្សែកោងរលាយនៃ apalene (NM_033207, F: GCCATGGAAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGAATCTCCTAGG) ។
C. ក្រាហ្វពង្រីក និងខ្សែកោងរលាយនៃ ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG)។
ឃ. ក្រាហ្វពង្រីក និងខ្សែកោងរលាយនៃ GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGAATCTTC)។
E. Cqs បានកត់ត្រានៅសីតុណ្ហភាព annealing ផ្សេងគ្នា ដែលបង្ហាញពីភាពខុសគ្នានៃ Cq ដែលបានកត់ត្រានៅក្រោមជម្រាលសីតុណ្ហភាព 7C ។
P 6 បង្ហាញពីលទ្ធផលធម្មតានៃការធ្វើតេស្តដែលមិនចង់បាន ដែល qPCR ត្រូវបានអនុវត្តដោយប្រើជម្រាល Tas រវាង 59C និង 67C (P 6a) ដោយប្រើ primers សម្រាប់ហ្សែនជាក់លាក់នៃខួរក្បាលរបស់មនុស្សចំនួនបី។
វាអាចត្រូវបានគេមើលឃើញពីក្រាហ្វពង្រីកដែលថា primers Opalin គឺនៅឆ្ងាយពីឧត្តមគតិ ដោយសារតែជួរ Ta ដ៏ល្អប្រសើររបស់ពួកគេគឺតូចចង្អៀតណាស់ (រូបភាព 6b) នោះគឺ Cqs ត្រូវបានបែកខ្ញែកយ៉ាងទូលំទូលាយ ដែលបណ្តាលឱ្យ Cqs ត្រូវបានគេប្រៀបធៀបយ៉ាងខ្លាំងទៅនឹង Cqs ទាបបំផុត។
វិធីសាស្ត្ររាវរកនេះមិនស្ថិតស្ថេរ ហើយអាចនាំទៅរកការពង្រីកបានល្អបំផុត។ដូច្នេះ primers គូនេះគួរតែត្រូវបានរចនាឡើងវិញ។លើសពីនេះ ការវិភាគខ្សែកោងរលាយ (inset) បង្ហាញថា ភាពជាក់លាក់នៃវិធីសាស្ត្ររាវរកនេះក៏អាចមានបញ្ហាដែរ ព្រោះខ្សែកោងរលាយនៃ Ta នីមួយៗមានភាពខុសគ្នា។
វិធីសាស្ត្ររាវរក ACSBG1 ដែលបង្ហាញក្នុង P 6c គឺរឹងមាំជាងវិធីសាស្ត្ររាវរក Opalin ខាងលើ ប៉ុន្តែវានៅតែឆ្ងាយពីឧត្តមគតិ ហើយវាទំនងជាអាចធ្វើឱ្យប្រសើរឡើងបាន។
ទោះជាយ៉ាងណាក៏ដោយ យើងសង្កត់ធ្ងន់ថាមិនមានការតភ្ជាប់ចាំបាច់រវាងភាពរឹងមាំ និងភាពជាក់លាក់នោះទេ ព្រោះខ្សែកោងរលាយដែលផលិតដោយវិធីសាស្ត្ររាវរកនេះបង្ហាញពីតម្លៃកំពូលដូចគ្នានៅក្នុង Tas (inset) ទាំងអស់។
ម៉្យាងវិញទៀត ការធ្វើតេស្តភាពរឹងមាំគឺមានភាពអត់ធ្មត់ច្រើន ដោយផលិត Cqs ស្រដៀងគ្នានៅក្នុងជួរដ៏ធំទូលាយនៃ Tas ដូចនៅក្នុងការធ្វើតេស្ត GFAP ដែលបានបង្ហាញនៅក្នុង P 6d ។
ភាពខុសគ្នានៃ Cqs ដែលទទួលបានក្នុងជួរ 8 អង្សាសេដូចគ្នាគឺតិចជាង 1 ហើយខ្សែកោងរលាយ (inset) បញ្ជាក់ពីលក្ខណៈរាវរកនៅក្នុងជួរសីតុណ្ហភាពនេះ។គួរកត់សម្គាល់ថា Tas ដែលបានគណនា និងជួរ Ta ពិតប្រាកដអាចខុសគ្នាខ្លាំង។
មានគោលការណ៍ណែនាំជាច្រើនដែលត្រូវបានរចនាឡើងដើម្បីជួយអ្នកស្រាវជ្រាវក្នុងការរចនាថ្នាំ primers ប្រកបដោយប្រសិទ្ធភាព ដែលភាគច្រើនគឺផ្អែកលើច្បាប់ដែលបានបង្កើតជាយូរមកហើយ ហើយការយកចិត្តទុកដាក់ជាច្រើនត្រូវបានបង់ទៅឱ្យ 3′ ចុងនៃ primers ។ជារឿយៗវាត្រូវបានណែនាំឱ្យបញ្ចូល G ឬ C នៅចុង 3' និងមូលដ្ឋាន G ឬ C ពីរ (GC clamp) ប៉ុន្តែមិនលើសពីពីរនៃមូលដ្ឋាន 5 ចុងក្រោយ។
នៅក្នុងការអនុវត្ត ច្បាប់ទាំងនេះអាចណែនាំអ្នកស្រាវជ្រាវ ប៉ុន្តែពួកគេមិនចាំបាច់ត្រឹមត្រូវក្នុងគ្រប់កាលៈទេសៈទាំងអស់នោះទេ។
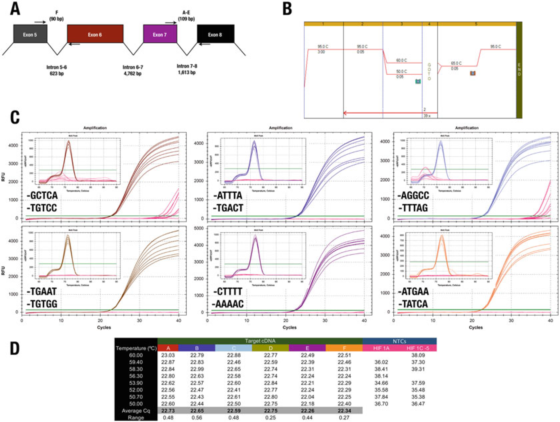
P៧ |ចុង 3′ នៃ primer មានឥទ្ធិពលតិចតួចលើភាពជាក់លាក់ឬប្រសិទ្ធភាព។
A. ទីតាំងនៃ primers សម្រាប់ហ្សែន HIF-1α (NM_181054.2) របស់មនុស្ស។
ខ. ប្រើ Agilent Brilliant III SYBR ស្រាម្តាយបៃតង (លេខឆ្មា 600882) ដើម្បីពង្រីកធាតុសាកល្បងចំនួនប្រាំមួយ។
គ. ក្រាហ្វពង្រីក និងខ្សែកោងរលាយដែលកត់ត្រាដោយឧបករណ៍ CFX qPCR របស់ Bio-Rad និង 3′ primers ។NTCs ត្រូវបានបង្ហាញជាពណ៌ក្រហម។
ឃ. កំណត់ត្រា Cqs នៃធាតុសាកល្បងនីមួយៗ
ឧទាហរណ៍ លទ្ធផលនៅក្នុង P 7 ផ្ទុយនឹងច្បាប់ 3′។ការរចនាទាំងអស់បង្កើតលទ្ធផលដូចគ្នាជាមូលដ្ឋាន ដោយមានតែការផ្សំបឋមពីរប៉ុណ្ណោះ ដែលនាំទៅដល់ការពង្រីកមិនជាក់លាក់នៅក្នុង NTC ។
ទោះយ៉ាងណាក៏ដោយ យើងមិនអាចគាំទ្រឥទ្ធិពលនៃឈុត GC បានទេ ព្រោះក្នុងករណីនេះ ការប្រើ A ឬ T ជាអតិបរមា 30 មូលដ្ឋានមិនកាត់បន្ថយភាពជាក់លាក់ទេ។
តេស្ត C ជាកន្លែងដែល F primer បញ្ចប់នៅក្នុង GGCC បានធ្វើកំណត់ត្រា Cqs នៅក្នុង NTCs ដោយបង្ហាញថាមនុស្សម្នាក់ប្រហែលជាចង់ជៀសវាងនូវលំដាប់ទាំងនេះនៅ 30-end។យើងសង្កត់ធ្ងន់ថាមធ្យោបាយតែមួយគត់ដើម្បីកំណត់លំដាប់ 3'end ដ៏ល្អបំផុតនៃគូ primer គឺដើម្បីវាយតម្លៃបេក្ខជនបឋមមួយចំនួនដោយពិសោធន៍។
ប្រសិទ្ធភាពពង្រីក
សំខាន់ ទោះបីជាការរកឃើញ PCR មិនជាក់លាក់មិនអាចក្លាយជាជាក់លាក់ក៏ដោយ ប្រសិទ្ធភាពនៃការពង្រីកអាចត្រូវបានកែតម្រូវ និងពង្រីកអតិបរមាតាមវិធីផ្សេងៗគ្នាជាច្រើន ដោយការផ្លាស់ប្តូរអង់ស៊ីម ស្រាម្តាយ សារធាតុបន្ថែម និងលក្ខខណ្ឌនៃការជិះកង់។
ដើម្បីវាយតម្លៃប្រសិទ្ធភាពនៃការរកឃើញ PCR វាជាការល្អបំផុតក្នុងការប្រើការរំលាយសៀរៀល 10 ឬ 5 ដងនៃអាស៊ីត nucleic គោលដៅ នោះគឺជា "វិធីសាស្ត្រខ្សែកោងស្តង់ដារ" ។
ប្រសិនបើ PCR amplicons ឬគោលដៅ DNA សំយោគត្រូវបានប្រើដើម្បីបង្កើតខ្សែកោងស្តង់ដារ ការបន្ថយសៀរៀលនៃគោលដៅទាំងនេះគួរតែត្រូវបានលាយបញ្ចូលគ្នាជាមួយនឹងចំនួនថេរនៃ DNA ផ្ទៃខាងក្រោយ (ដូចជា DNA ហ្សែន)។
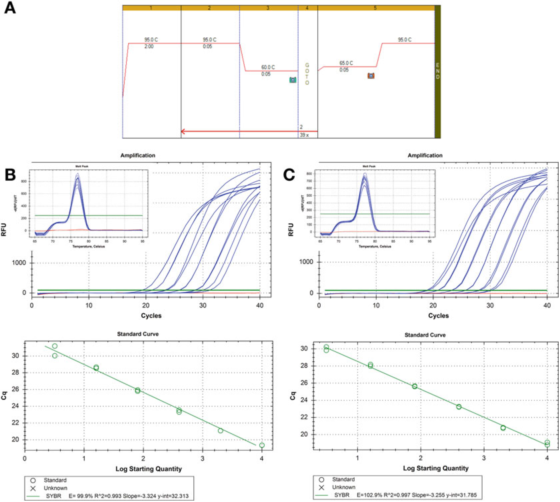
P៨ |ខ្សែកោង Dilution ដើម្បីវាយតម្លៃប្រសិទ្ធភាពនៃ PCR ។
A. ប្រើ primers សម្រាប់ HIF-1: F: AAGAACTTTTAGGCCGCTCA និង R: TGTCCTGTGGTGACTTGTCC និង Agilent's Brilliant III SYBR Green mastermix (លេខកាតាឡុក 600882) សម្រាប់ PCR និងលក្ខខណ្ឌខ្សែកោងរលាយ។
ខ. 100 ng RNA ត្រូវបានចម្លងបញ្ច្រាស ពនឺ 2 ដង ហើយសំណាក cDNA ដែលត្រូវបានពនឺតាមលំដាប់លំដោយត្រូវបានពនឺ 5 ដងទៅ 1 ng DNA ហ្សែនរបស់មនុស្ស។ខ្សែកោងរលាយត្រូវបានបង្ហាញនៅក្នុង inset ។
C. ប្រតិកម្ម RT ការពនរ និងការពនរសៀរៀលត្រូវបានធ្វើម្តងទៀតសម្រាប់គំរូ cDNA ទីពីរ ហើយលទ្ធផលគឺស្រដៀងគ្នា។
P 8 បង្ហាញពីខ្សែកោងស្ដង់ដារពីរ ដោយប្រើវិធីសាស្ត្ររាវរកដូចគ្នានៅលើគំរូ cDNA ពីរផ្សេងគ្នា លទ្ធផលគឺប្រសិទ្ធភាពដូចគ្នាប្រហែល 100% ហើយតម្លៃ R2 ក៏ស្រដៀងគ្នាដែរ ពោលគឺកម្រិតសមរវាងទិន្នន័យពិសោធន៍ និងបន្ទាត់តំរែតំរង់ ឬទិន្នន័យកម្រិតលីនេអ៊ែរ។
ខ្សែកោងស្ដង់ដារទាំងពីរអាចប្រៀបធៀបបាន ប៉ុន្តែមិនដូចគ្នាបេះបិទទេ។ប្រសិនបើគោលបំណងគឺដើម្បីកំណត់បរិមាណគោលដៅឲ្យបានត្រឹមត្រូវ វាត្រូវតែត្រូវបានកត់សម្គាល់ថាវាមិនអាចទទួលយកបានទេក្នុងការផ្តល់ការគណនាលេខចម្លងដោយមិនពន្យល់ពីភាពមិនច្បាស់លាស់។
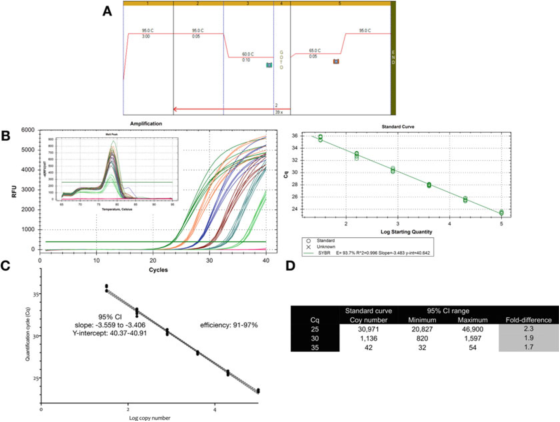
P៩ |ភាពមិនច្បាស់លាស់នៃការវាស់វែងដែលទាក់ទងនឹងបរិមាណដោយប្រើខ្សែកោងស្តង់ដារ.
ក. ប្រើ primers សម្រាប់ GAPDH (NM_002046) ដើម្បីអនុវត្តលក្ខខណ្ឌ PCR និងរលាយ។F: ACAGTTGCCATGTAGACC និង R: TAACTGGTTGAGCACAGG និង ល្បាយមេ Sensifast SYBR របស់ Bioline (លេខកាតាឡុក BIO-98050)។
ខ. គំនូសតាងពង្រីក ខ្សែកោងរលាយ និងខ្សែកោងស្ដង់ដារដែលបានកត់ត្រាជាមួយនឹងឧបករណ៍ CFX qPCR របស់ Bio-Rad ។
គ. ក្រាហ្វខ្សែកោងស្តង់ដារ និងចន្លោះពេលទំនុកចិត្ត 95% (CI)។
D. លេខចម្លង និងចន្លោះពេលទំនុកចិត្ត 95% នៃតម្លៃ Cq ទាំងបីដែលបានមកពីខ្សែកោង dilution ។
P 9 បង្ហាញថាសម្រាប់ការធ្វើតេស្តដែលបង្កើនប្រសិទ្ធភាព ភាពប្រែប្រួលនៃខ្សែកោងស្តង់ដារតែមួយគឺប្រហែល 2 ដង (ចន្លោះពេលទំនុកចិត្ត 95% អប្បបរមាដល់អតិបរមា) ដែលអាចជាការប្រែប្រួលតូចបំផុតដែលអាចរំពឹងទុកបាន។
ផលិតផលដែលពាក់ព័ន្ធ៖
ឧបករណ៍ PCR ផ្ទាល់របស់កណ្តុរកន្ទុយ
ឧបករណ៍ PCR ដោយផ្ទាល់ជាលិកាសត្វ
ពេលវេលាប្រកាស៖ ថ្ងៃទី ៣០ ខែកញ្ញា ឆ្នាំ ២០២១